科學家研發出單細胞和空間轉錄組中環形RNA深度學習算法
軍工資源網 2024年02月23日環形RNA是一類廣泛表達的非編碼RNA,有較高的細胞類型及組織表達特異性,在器官發育及腫瘤發生等過程中起著重要調控作用。環形RNA由3‘端的受體位點和5’端的供體位點共價連接形成,這一過程被稱為反向剪接,然而由于缺少poly(A)尾,環形RNA無法被經過polyA富集等轉錄組建庫方式有效捕獲。因此,環形RNA的表達信息在絕大部分單細胞以及空間轉錄組數據中缺失。為了獲得豐富的單細胞及空間水平的環形RNA表達信息,需要研發新型環形RNA表達預測算法。
中國科學院北京基因組研究所研究員高遠團隊與北京生命科學研究院研究員趙方慶團隊聯合研發了深度學習模型CIRI-deep,以準確預測不同樣本間的差異剪接環形RNA。該模型從環形RNA調控機制角度出發,整合了3527個環形RNA特異的順式元件以及1499個樣本特異的反式因子作為輸入特征,且不依賴于傳統的反向剪接信號識別,可以在任意轉錄組樣本間預測差異剪接的環形RNA。評估結果表明,CIRI-deep可以實現多種轉錄組測序數據中差異剪接環形RNA的可靠預測,并在單細胞及空間水平實現細胞類型特異環形RNA的準確解析,具有廣泛的應用場景。
研究團隊從397個深度測序的全轉錄組樣本中識別了超過2500萬高度可信的環形RNA差異剪接事件。由于這些剪接事件覆蓋了25個人體組織,使用它們作為訓練集,使CIRI-deep具有良好的泛化性能。CIRI-deep在測試數據集上的AUROC值達到0.906,并且可以準確預測來自非訓練集病理條件與正常樣本間的環形RNA差異剪接。此外,在低深度測序的轉錄本中,CIRI-deep對差異環形RNA的預測效果優于基于reads數的統計檢驗方法。
為了解釋CIRI-deep的預測原理,研究團隊研發了深度學習可解釋性分析框架Adapted Integrated Gradient,以量化分析組織特異環形RNA的調控因素及其貢獻。結果表明,相較于基因序列結構等順式元件,RNA結合蛋白等反式因子的表達水平對預測準確性的貢獻更大,且具有更強的組織特異性。該分析框架驗證了已知的環形RNA剪接的調控因素,如剪接位點、內含子區域的Alu元件、FUS蛋白的表達等,也提示了之前未發現的潛在調控因子如NOVA2、KHDRBS3等對環形RNA剪接的影響。
為了從polyA富集測序的單細胞以及空間轉錄組數據中挖掘環形RNA表達水平差異,研究團隊進一步利用polyA數據訓練了CIRI-deepA模型。結果表明,CIRI-deepA的預測表現大幅超過直接使用polyA數據推斷差異剪接環形RNA的效果。在腦膠質瘤數據集上應用CIRI-deepA,表明該模型可有效預測腫瘤細胞群體和健康細胞群體之間的差異剪接環形RNA。研究團隊也將CIRI-deepA應用到了10X單細胞數據集上,準確預測了不同細胞群體的特異高表達環形RNA。另外,在空間轉錄組數據中,CIRI-deepA可用于預測空間區域特異高表達的環形RNA,并實現對環形RNA表達進行空間區域水平的可視化。利用CIRI-deepA預測的高度特異表達環形RNA,可進一步解析不同區域的細胞類型構成。
CIRI-deep模型可有效用于各轉錄組樣本間推斷差異剪接環形RNA,拓展了環形RNA的研究范圍,為環形RNA研究提供了新的高效分析方法。同時,CIRI-deepA模型可以提供單細胞及空間水平環形RNA的有效解析,為挖掘細胞類型特異的環形RNA標志物提供了重要的方法學工具。
2月2日,相關成果以CIRI-Deep Enables Single-Cell and Spatial Transcriptomic Analysis of Circular RNAs with Deep Learning為題,發表在《先進科學》(Advanced Science)上。研究工作得到國家重點研發計劃、國家自然科學基金等的支持。
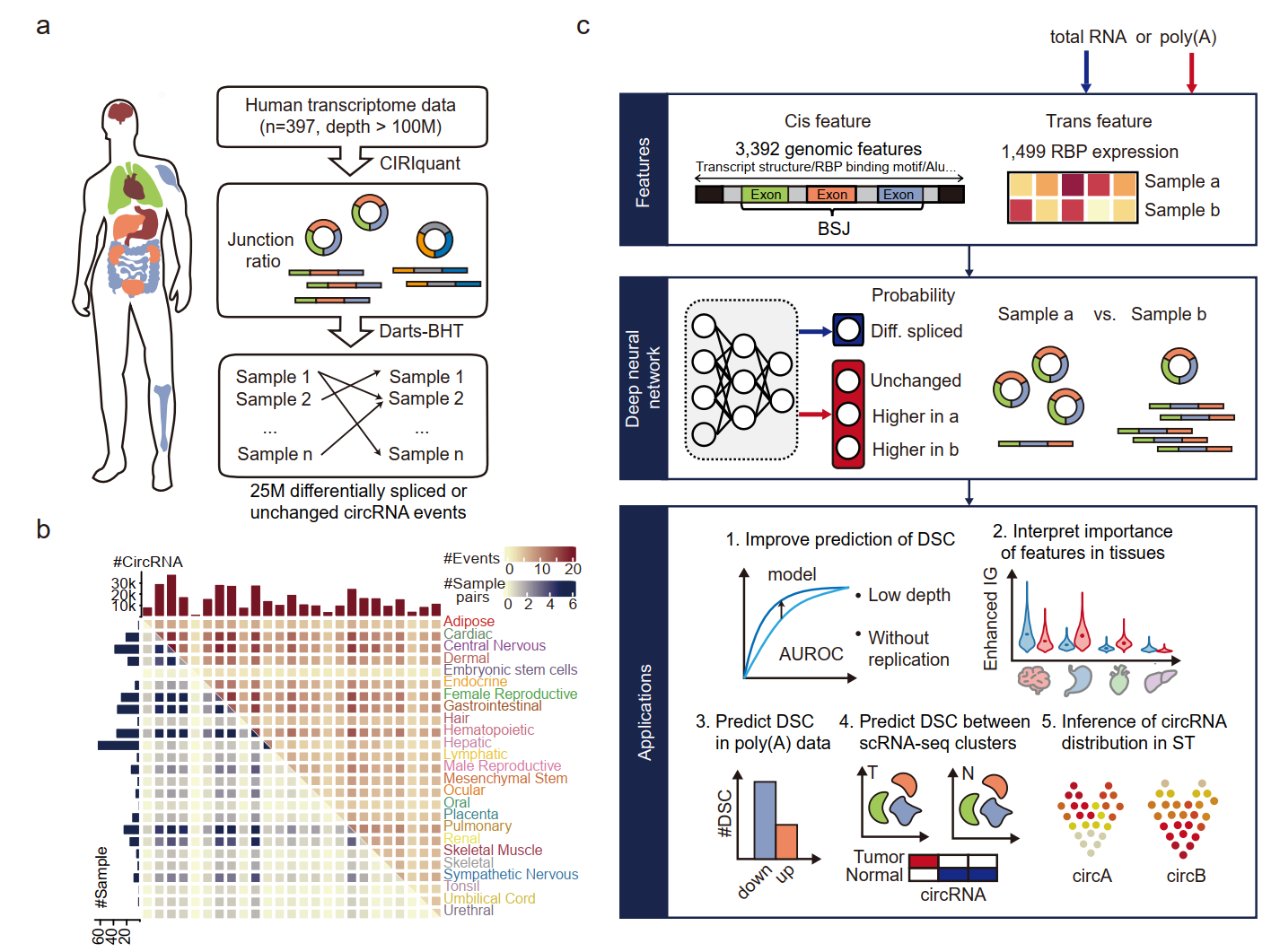
基于深度學習的環形RNA差異剪接預測算法CIRI-deep
